 |
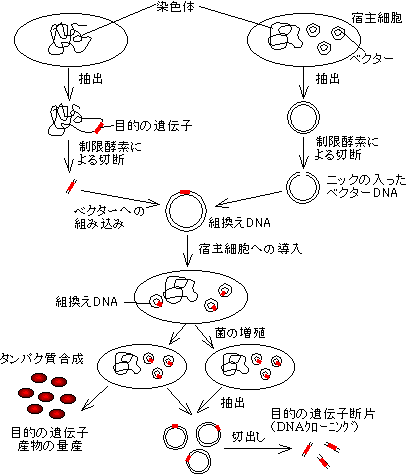
《基本的な手順》 |
| 1. 目的のDNA断片の調製 2. 組換え体DNAの作成 3. 組換え体を宿主細胞に導入 4. 組換え体を含む細胞の検出と選別 |
|
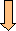 |
|
| DNAのクローニング(cloning) 宿主細胞からの組換え体DNAの抽出 ↓ 目的DNAの切り出し ↓ DNAの1次構造解析 |
|
| 組換え体を含む細胞の増殖 ↓ 有用物質の生産 |
|


|
遺伝子工学の道具 制限酵素 組み換え体の作成 クローニングベクター 組み換え体の導入と検出 組み換え体の発現 PCR法 |
遺伝子DNAを細胞から取り出し、人工的な操作を加えたり、それを利用して遺伝子産物(タンパク質)を細胞につくらせる技術を遺伝子工学(gene technology, genetic engineering)、遺伝子操作(gene manipulation)、遺伝子組み換え技術(recombinant DNA technique)などと呼ぶ。
遺伝子工学にはいくつかの道具が必要である。1970年に相次いで発見された制限酵素と逆転写酵素は、遺伝子工学を現実のものとした。制限酵素はDNAの特定の塩基配列を認識して切断するため、目的遺伝子の切り出しに欠かせない。逆転写酵素はmRNAに相補的な放射能標識したcDNAを作るのに用いられる。cDNAは目的遺伝子の検出やそれ自体のクローニングに利用される。リガーゼはDNAの切れ目をつなぐ酵素で、 組み換え体(recombinant)を作成するのに用いられる。遺伝子を組み込む相手として用いられるDNAをベクターと呼ぶ。現在広く用いられているベクターとしては、ファージ、動植物ウィルスおよびプラスミドなどがある。プラスミドは多くのバクテリアに存在する小型の核外遺伝子で、組み換え体を選別するためのマーカー遺伝子や複製開始点を持つ。この他、長鎖のDNAを組み込むため、lファージとプラスミドを基に人工的に作られたコスミドや酵母の人工染色体などが用いられる。組み換え体を取り込ませ増やすためには、大腸菌、酵母などの宿主細胞が必要である。
組み換え体を宿主細胞に入れ、目的遺伝子を増やしてDNA断片を量的に得る操作をDNAのクローニングと呼ぶ。その塩基配列を決定することによって、遺伝子の構造や遺伝子の制御の仕組みを知ることが可能となった。一方、組み換え体を用いて、目的のタンパク質を作らせることも出来る。ヒトのタンパク質を微生物で作らせる場合、目的の遺伝子をリボソーム結合部位(SD配列)の後に組み込む必要がある。このために開発されたベクターを特に発現ベクターと呼ぶ。近年、DNAの新しい増殖法としてPCR法が開発され、微量の試料からでもDNAをクローニングできるようになった。
《基本的な手順》 1. 目的のDNA断片の調製
2. 組換え体DNAの作成
3. 組換え体を宿主細胞に導入
4. 組換え体を含む細胞の検出と選別DNAのクローニング(cloning)
宿主細胞からの組換え体DNAの抽出
↓
目的DNAの切り出し
↓
DNAの1次構造解析組換え体を含む細胞の増殖
↓
有用物質の生産
  
|
|
 cDNAの調製
cDNAの調製
細胞で発現している遺伝子産物の構造を調べる目的で,mRNAのヌクレオチド配列を決定することが行われる。mRNAは3’末端にポリ(A)鎖をもつので,オリゴ(T)をプライマーとして逆転写酵素を作用させると,mRNAに相補的なDNA鎖が合成できる。アルカリ処理でmRNAを分解後,生じた1本鎖DNAを鋳型として逆転写酵素またはDNAポリメラーゼで2本鎖にし,クローニングに供される。
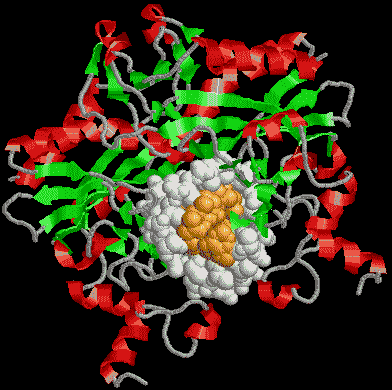
[逆転写酵素を用いるcDNAの調製]
[逆転写酵素の立体構造]レトロウィルスHIV-1の酵素で,二量体である。
阻害剤であるdipyridodiazepinone nevirapineが分子左下方に結合している。
  
|
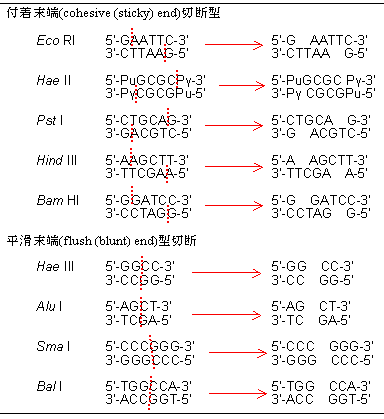
DNAの特定の塩基配列を認識して切断する酵素を制限酵素(restriction enzyme)という。特に、II型の制限酵素は遺伝子工学においてDNAの断片化に頻繁に用いられる。(遺伝子工学の道具)
 I 型制限酵素
I 型制限酵素
 II 型制限酵素
II 型制限酵素
| 代表的なII型の制限酵素とその認識部位 縦線が切断個所 |
|
 |
 |
| [DNA鎖に結合した制限酵素] | |
| EcoRV がDNA鎖を切断している様子を DNA鎖の真上方向からから見た図。 EcoRVは次のような回文構造 GATATC CTATAG の真中を切断する。 |
 III型制限酵素
III型制限酵素
|
《目的DNA断片を調製する》
** 遺伝子ライブラリやcDNAライブラリが利用できる場合は、それらを用いる。
DNA mRNA* ↓制限酵素 ↓ 逆転写酵素 たくさんのDNA断片 cDNAの混合物 ↓ 電気泳動 ↓ PCR法で増やす ↓ ブロッティング ↓ 電気泳動 ↓ ハイブリダイゼーション ↓ バンドを検出 ↓ オートラジオグラフィー ↓ ゲルから抽出 DNAの断片** DNAの断片**
 付着末端を生じる制限酵素でDNA断片が切り出せた場合
付着末端を生じる制限酵素でDNA断片が切り出せた場合
 制限酵素法
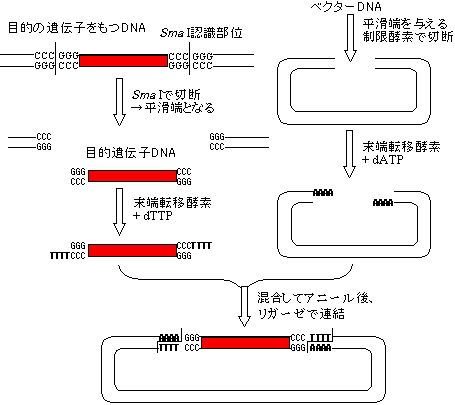
制限酵素法 平滑末端を生じる制限酵素でDNA断片が切り出せた場合
平滑末端を生じる制限酵素でDNA断片が切り出せた場合
 ホモポリマー法
ホモポリマー法 T4リガーゼ結合法
T4リガーゼ結合法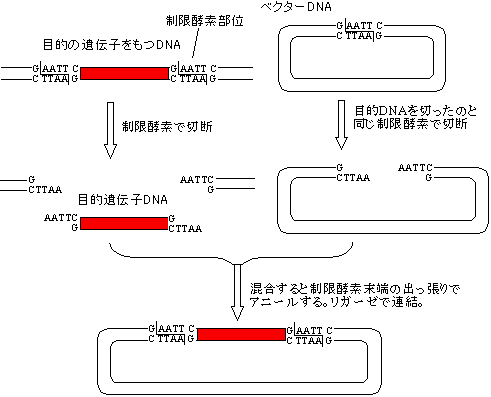 |
||
| [制限酵素切断末端をのりしろにする方法] | ||
| 付着端を生じる制限酵素でDNA断片が切り出せた場合はベクターも同じ制限酵素 で切り、切断片をのりしろにしてアニールさせる。切れ目をリガーゼでつなぐ。 |
||
|
||
 |
||
| [ホモポリマーを結合させてのりしろにする方法] | ||
| 平滑端のDNA断片しか得られなかった場合は、末端転移酵素を用いて, DNA断片にオリゴ(T)、ベクターにオリゴ(A)を結合させてのりしろにする。 |
||
|
||
 (cloning vectors) (cloning vectors) |
  |
 プラスミド系ベクター
プラスミド系ベクター
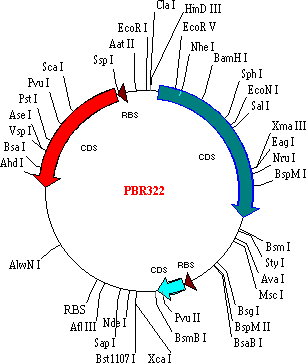
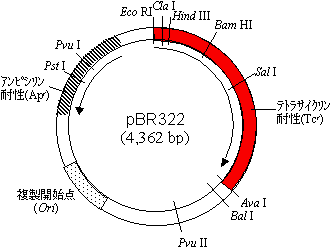  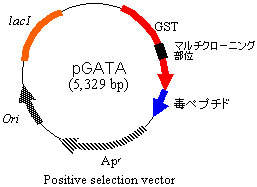 [いろいろなプラスミドベクター] [いろいろなプラスミドベクター] |
| 矢印はマーカー遺伝子のセンス鎖の方向。細い線は制限酵素の作用部位。 lacI : lacリプレッサー、 lacZ : b-ガラクトシダーゼ、 GST: グルタチオン-S-トランスフェラーゼ。 マルチクローニング部位: 種々の制限酵素で切断可能な配列(例)。 |
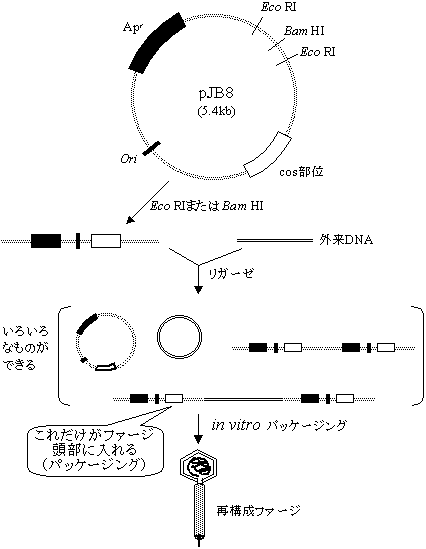
 ファージベクター
ファージベクター
[lファージ遺伝子]
赤はファージの感染に不要な部分。ファージDNAが頭部に収納されるためにはcos部位と呼ばれる12塩基の相補的な2つの1本鎖配列が両端にあり、これらが36〜51kb離れていることが必要である。ファージタンパク質とDNAを試験管内で混ぜるだけで、成熟ファージ粒子が出来あがる(in vitro packaging)。 [lファージ粒子の形成]
| 遺伝子ライブラリやcDNAライブラリの作成 in vitro packagingでファージ頭部に入りうるDNAの大きさには制限がある(野生ファージゲノムの78〜105%の長さ)。1つの生物の遺伝子を制限酵素で分解して得られたDNA断片をファージに組み込むと、ほぼ一定の長さのものだけを詰め込むことができることになる。このようなDNAの集合物を遺伝子ライブラリという。ファージに組み込まれたDNAは安定に保存できるし、増やすのも容易なので、多くの人が迅速に利用できる。 細胞の全mRNAをcDNAに変え、同様にパッケージしたものをcDNAライブラリと呼ぶ。 |
 コスミド・ベクター
コスミド・ベクター
 酵母人工染色体(YAC, yeast artificial chromosome)
酵母人工染色体(YAC, yeast artificial chromosome)
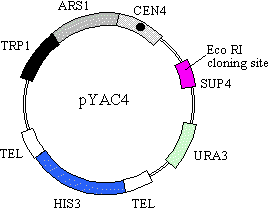
| 《一言》 数Mbpも離れた遺伝子を検索するために染色体歩行を行う場合、YACベクターはサイズが大きいクローンを得ることができるので大変有利である。 YACベクターを構築する場合、プラスミドpBR322のアンピシリン耐性遺伝子と複製開始点を組み込んだシャトルベクターをつくり、大腸菌で大量に調製する。ベクター自体の大きさは約5〜20kbpである。 |
3種のベクターの比較 目 的 プラスミド ファージ コスミド 10kb以上のDNAのクローニング − + + 遺伝子ライブラリの作成 − + + cDNAライブラリの作成 + − − 小さな(<10kb)DNAのサブクローニング + − − 大腸菌での遺伝子発現 + − −
  
|
 宿主細胞への導入
宿主細胞への導入
大腸菌へのプラスミドの取り込み
- 菌を氷冷下、CaCl2で処理すると、DNAを取り込むようになる(コンピテントな状態という)。
ベクターとしてファージを利用する
- 感染によって取り込ませる。菌に入ったファージは隣の菌に次々と感染し、溶菌班(プラーク)を形成。
動物細胞へのDNAの取り込み(トランスフェクションという)
- カルシウム−リン酸共沈法(細胞の食作用を利用)
その他、種々の方法がある。
[組換え体の導入法]
 目的遺伝子を受け取った細胞の選択的検出
目的遺伝子を受け取った細胞の選択的検出
挿入失活法
- pBRプラスミド: テトラサイクリン(Tc)とアンピシリン(Ap)耐性マーカー遺伝子が存在
2段階選別法。
[マーカー遺伝子を利用した組み換え大腸菌の選別]直接選別法
- b-ガラクトシダーゼのを組み込んだpUC系ベクターで、目的DNA断片の挿入失活を利用する。基質X-galを作用させると、b-ガラクトシダーゼが作られていれば(lacZ'遺伝子が分断されていなければ)X-galが分解されて青色を呈し、青いコロニーを生じる。
lacZ'遺伝子の途中には遺伝子を組み込むためのマルチクローニング部位がある。もし,ここに目的DNA断片が挿入されていれば、b-ガラクトシダーゼを作れない。従って,組換え体を取り込んだ細胞はX-galを分解できないため白いコロニーを生じるので,これを選別すればよい。
[直接選別法による組み換え大腸菌の選別]
コロニー(colony): 細胞の集落のこと
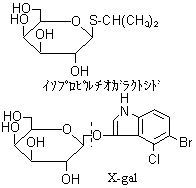
- IPTG(イソプロピルチオガラクトシド):
- 乳糖(ラクトース)と同様、lac リプレッサータンパク質に結合してリプレッサー活性を抑え、lac Z(b-ガラクトシダーゼ)遺伝子の転写を開始させる。しかし、乳糖と異なりb-ガラクトシダーゼで分解されないので、持続的に作用を発揮する(左図)。
- x-gal:
- ガラクトースの誘導体で,b-ガラクトシダーゼで分解されると青色の色素を遊離する(左図)。
コロニーハイブリダイゼーション法
[コロニーハイブリダイゼーション法による組み換え体の検出]
 DNAのクローニング
DNAのクローニング
  
|
目的遺伝子の入った細胞を使ってタンパク質を生産したいが?
このためには、宿主細胞のプロモーター、リボソーム結合(Shine-Dalgarno)配列を配置したベクターの適切な位置に目的DNAを組み込む必要がある。
[目的遺伝子のタンパク質の合成]
最初の頃は、大腸菌プラスミドのラクトースオペロンのb-ガラクトシダーゼ遺伝子の途中にDNA断片を挿入して、b-ガラクトシダーゼN端部との融合タンパクとして多くのタンパク質がつくられた。
[合成遺伝子を用いるタンパク質の生産]
現在では、タンパク質合成能を持つベクター(発現ベクター)が用意されている。lac(ラクトースオペロン), trp(トリプトファンオペロン), tac(lacとtrpオペロンを融合したもの)のプロモーターを組み込んだ発現プラスミド(大腸菌)、バキュロウィルス由来のベクター(昆虫細胞中で糖タンパク質も作れる)、酵母の発現ベクターなどがよく使われる。
[発現プラスミドの構成]
プラスミドpU18/19はこの目的に利用可能。
マルチクローニング部位の前にプロテアーゼ認識
部位を入れておくと、後で遺伝子産物を切り出せる。
 Polymerase Chain Reaction法 Polymerase Chain Reaction法 |
 |
1985年、Mullisによって考案された画期的なDNA増幅法である。1988年、耐熱性のDNAポリメラーゼ(Taq DNAポリメラーゼ)が導入され、実用化された。
PCR法は次の3つの段階からなり,このサイクルを(1)〜(3)を20回ほど繰り返すことにより、短時間にかつ簡便にDNA断片を百万倍以上に増幅できる。
(1) 2本鎖DNAの熱変性->1本鎖へ解離
(2) 2つのプライマーのアニーリング
(3) DNAポリメラーゼによるDNA鎖の延長
増幅するDNAの範囲は,設計したプライマーの配列に依存する。下の例ではプライマーAとBで挟まれた範囲だけが増幅される。<PCRサイクル2>で初めて目的のDNA断片が1組生成し(二重丸),以後は、nサイクルで、2n−n−1組になる。
n=10→1,013組, n=20→1,048,555組
プライマー
鋳型DNA
PCRサイクル1 1. 熱変性
2. プライマー結合
3. プライマー伸長反応PCRサイクル2 1〜3
PCRサイクル3 1〜3
PCRサイクルの繰り返し [PCR法の原理]
| 増幅したい標的DNA断片の配列をS+とS-とする。 | |
| (S+) 5' ATCAACTAATA-------ACGACGAGGATG 3' (S-) 3' TAGTTGATTAT-------TGCTGCTCCTAC 5' |
|
| この2本鎖の3'末端部分に相補的な短いオリゴヌクレオチド(プライマー)を準備する(合成品を使う)。 | |
| プライマーA: 3' CTCCTAC 5' (青部分の相補鎖) プライマーB: 5' ATCAACT 3' (赤部分の相補鎖) |
|
| 標的配列を含むDNA, プライマーAとB, 4種のdNTP, 耐熱性のDNAポリメラーゼを混合し,反応を開始する。反応といっても,温度を上げ下げするだけ! | |
<サイクル1> 5' ---
3' ---ATCAACTAATA-------ACGACGAGGATG
TAGTTGATTAT-------TGCTGCTCCTAC--- 3'
--- 5'標的配列を含む
DNA1. 熱変性(96℃,15秒)
2. プライマーのアニーリング(55℃, 30秒)
5' --- ATCAACTAATA-------ACGACGAGGATG --- 3' ● CTCCTAC
(プライマーA) 3' --- TAGTTGATTAT-------TGCTGCTCCTAC --- 5' ● ATCAACT (プライマーB) 3. DNAが鎖を伸長(72℃, 90秒)
●から 5' ---
3' ---ATCAACTAATA-------ACGACGAGGATG
TAGTTGATTAT-------TGCTGCTCCTAC--- 3'
5'▲
■●から 3' ---
5'TAGTTGATTAT-------TGCTGCTCCTAC
ATCAACTAATA-------ACGACGAGGATG--- 5'
--- 3'▲
■<サイクル2> 1〜3
▲から 5' ---
3' ---ATCAACTAATA-------ACGACGAGGATG
TAGTTGATTAT-------TGCTGCTCCTAC--- 3'
5'■から 3' ---
5'TAGTTGATTAT-------TGCTGCTCCTAC
ATCAACTAATA-------ACGACGAGGATG5'
3'(S+) ▲から 3' ---
5'TAGTTGATTAT-------TGCTGCTCCTAC
ATCAACTAATA-------ACGACGAGGATG--- 5'
--- 3'■から 5'
3'ATCAACTAATA-------ACGACGAGGATG
TAGTTGATTAT-------TGCTGCTCCTAC--- 3'
5'(S-)

|