









|
�@mRNA�𒒌^�Ƃ��ă^���p�N����������i�K���|��(translation)�Ƃ����B�^���p�N���̐������ɂ�mRNA�ȊO�ɁA�g�����X�t�@�[RNA(tRNA)��{�\�[�����K�v�ł���B���j�זE�Ɛ^�j�זE�̖|��@�\�͑�ϗǂ����Ă���B�����ɂƂ��ďd�v�ȋ@�\�͐�������ĕۑ�����Ă������ʂƂ�����B
 (ribosome) (ribosome) |
  |
 |
|
||
|
|||
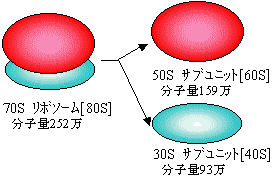
| [���j�זE�Ɛ^�j�זE�̃��{�\�[���̃T�u���j�b�g�\���ƍ\������] ���F�g���͐^�j�זE�̏ꍇ |
|||
![]() ���{�\�[���̎p
���{�\�[���̎p
�@�d�q�������łƂ炦��ꂽ������A�咰�ۂ̃��{�\�[���͉��̂悤�Ȋ�Ȍ`�����Ă���Ɛ��肳��Ă����B
�@�ŋ߁C���j�זE�̃��{�\�[���̗��̍\����X����͂ɂ���Č��肳�ꂽ�
 |
| [���j�זE�̃��{�\�[���̗��̍\��] |
| 30S�T�u���j�b�g�i���j��50S�T�u���j�b�g�i�E�j���������킹�Ă��鑤�ʐ}�B RNA�𔒂̃��{���ŁC�^���p�N���̃A�~�m�_�c�����X�̐F�̋��Ŏ���� |
 |
 |
||
| [���{�\�[��50S�T�u���j�b�g] ���ʁi30S�ƌ�������ʁj���猩���} |
[���{�\�[��30S�T�u���j�b�g] ���ʁi50S�ƌ�������ʁj���猩���} |
||
| �\���F | 23S rRNA(2904 nt)�C5S rRNA(120 nt)�@���q�� �v110�� 34��̃^���p�N���@���q�� �v49�� |
�\���F | 16S rRNA(1541 nt)�@���q�� 56�� 21��̃^���p�N���@���q�� �v37�� |
  
|
| GAAUAGGCGCCATTAGAUAUG GUU UGU UUU GCG.�c�cCAU UAA UAUAGCGAUUUU.�c�c | ||
| �J�n�R�h�� | �I�~�R�h�� | |
��`�Í��\(mRNA�̃R�h���j �@ �Q������ �@ U C A G �P
��
��
��U UUU Phe
UUC Phe
UUA Leu
UUG LeuUCU Ser
UCC Ser
UCA Ser
UCG SerUAU Tyr
UAC Tyr
UAA �I�[�J�[
UAG �A���o�[UGU Cys
UGC Cys
UGA �I�p�[��
UGG TrpU
C
A
G�R
��
��
��C CUU Leu
CUC Leu
CUA Leu
CUG LeuCCU Pro
CCC Pro
CCA Pro
CCG ProCAU His
CAC His
CAA Gln
CAG GlnCGU Arg
CGC Arg
CGA Arg
CGG ArgU
C
A
GA AUU Ile
AUC Ile
AUA Ile
AUG MetACU Thr
ACC Thr
ACA Thr
ACG ThrAAU Asn
AAC Asn
AAA Lys
AAG LysAGU Ser
AGC Ser
AGA Arg
AGG ArgU
C
A
GG GUU Val
GUC Val
GUA Val
GUG Val*GCU Ala
GCC Ala
GCA Ala
GCG AlaGAU Asp
GAC Asp
GAA Glu
GAG GluGGU Gly
GGC Gly
GGA Gly
GGG GlyU
C
A
G���͏I�~�R�h���B�@* ���j�����ł͊J�n�R�h���ƂȂ�
 �R�h���Έ�(codon bias)
�R�h���Έ�(codon bias)
�@�S�ẴR�h������l�Ɏg����킯�ł͂Ȃ��B�e�X�̃R�h���̎g�p�p�x�͐����ɂ���Ă��Ȃ�肪����B�I�~�R�h���iStop�j�̎g�p�p�x�̕肪���ɑ傫���B�R�h���o�C�A�X��PCR�̃v���C�}�[���f�U�C�����鎞�ɍl������K�v������
�R�h���ΈʂɊւ�������Əڍׂȏ��http://www.kazusa.or.jp/codon/
�q�g�Ƒ咰�ۂ̃R�h���g�p�p�x(%) �A�~�m�_ �R�h�� �q�g �咰�� Arg CGU
CGC
CGA
CGG
AGA
AGG8.4
19.6
11.0
20.6
20.0
19.837.3
38.1
6.6
10.2
4.9
2.9Ala GCU
GCC
GCA
GCG26.2
40.1
22.2
10.917.3
26.6
21.9
34.3Stop UAA
UAG
UGA28.5
20.8
50.761.7
8.0
30.3
  
|
 �@�咰�ۂ̃^���p�N������
�@�咰�ۂ̃^���p�N������
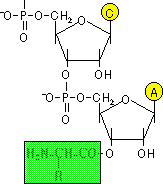
 �A�~�m�A�V��-tRNA�����y�f
�A�~�m�A�V��-tRNA�����y�f| �N���X�T�y�f(2'-OH) | �N���X�U�y�f(3'-OH) |
| Glu, Gln, Arg, Val, Ile, Leu, Met, Tyr, Trp | Gly, Ala, Pro, Ser, Thr, Asp, Asn, His, Lys |
 |
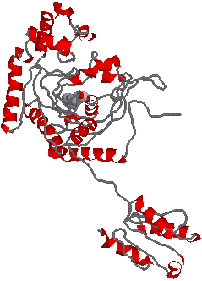 |
| [Met-tRNA�����y�f�̍\��] �N���X�T�y�f |
[His-tRNA�����y�f�̍\��] �N���X�U�y�f�i�y�f������His�������j |
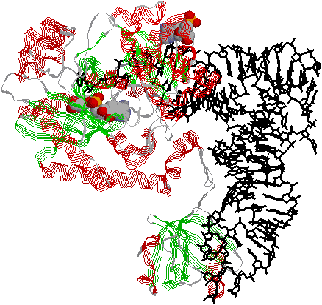 |
| [Asp-tRNA�����y�f��tRNAAsp������] |
| �}�̍�������Asp-AMP�ƌ�������Asp-tRNA�����y�f�B�E�̍����Ŏ����̂�Asp���ٓI��tRNA�BtRNA��3'���[��Asp�̂������Ɍ������Ă���̂�������B |
 + ATP + tRNA + ATP + tRNA |
 �A�~�m�A�V��-tRNA �����y�f |
 |
| tRNA��3' ���[ �i�A�~�m�_��2'-OH�� ��������ꍇ������j |
||
| [�A�~�m�A�V��-tRNA�̍����i�A�~�m�_�̊������j] | ||
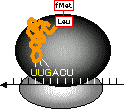
�J�n�R�h���ɑΉ�����J�ntRNA�itRNAfMet�j�ɂ͐悸���`�I�j�����������A���̃A�~�m��z���~��������ăz���~�����`�I�j���ɂȂ�B
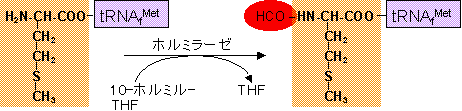
[���j�זE�̊J�ntRNA�̍���]
R17�t�@�[�WA�^���p�N��mRNA
16S rRNA 3' ���[[mRNA��Shine-Dalgarno�z��i���{�\�[���������ʁj��16S ��RNA 3'���[�̌���]
mRNA�̊J�n�R�h���ƊJ�ntRNA�̊ԂŌ�����������B
- IF-3�̗V���ɑ����C50S�T�u���j�b�g��30�r�T�u���j�b�g�Ɍ�������GTP�������������C���̕����̂Ɍ�������BIF-1��IF-2�������B����Formyl-���`�I�j�������������J�ntRNA�itRNAfMet�j����������i�^�j�����ł́A�J�ntRNA�̓��`�I�j�����^�ԁj�B����ɂ����50�r�T�u���j�b�g���t���A�����̂���������B
[�^���p�N���������̊J�n�i�咰�ہj�C�@70S �J�n�����̂̌`��]
- (3) �|���y�v�`�h���̉���
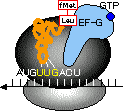
�����y�v�`�h����C���[�ɃA�~�m�_������R�i�K�����T�C�N���Ń^���p�N����������B- 1) mRNA�̃R�h���ɑΉ�����A�~�m�A�V��-tRNA-�������qTu-GTP�����̂����{�\�[����A������������B
2) A���ʂ̃A�~�m�A�V��-tRNA�̃A�~�m�P������tRNA�����j�u�����A�y�v�`�h�������`������i�y�v�`�h�]���j�B
3) �A�~�m�_������ċ�ƂȂ���tRNA��P���ʂ𗣂��B
4) A���ʂ̃y�v�`�W��-tRNA��mRNA����P���ʂɈړ����i�g�����X���P�[�V�����j�CGTP�����������������qG��A���ʂɌ�������B�ʔ������ƂɁCGTP�����������������qG�̗��̍\���̓A�~�m�A�V��-tRNA-�������qTu-GTP�����̂Ƃ�������ł���i���q�[���j�B
5)�������qG��GTP�������������ă��{�\�[���𗣂�C���̃T�C�N���ɓ���B- ���̃T�C�N�����J��Ԃ����ƂŁC�P�b�ԂɂR�`�T�̃A�~�m�_���A������Ă����B
[�咰�ۃ^���p�N���������ɂ�����y�v�`�h���̐L��]
�y�|���\�[���z
�|�J�n����ă��{�\�[�����ړ�����ƁA�ʂ̃��{�\�[�����J�n�ʒu�Ɍ������A�����̃��{�\�[����mRNA��Ő���Ȃ��ɂȂ��Ė|����s���B���̍\�����|���\�[��(polysome)�ƌĂԁB
(4) �������̏I���i���I���j
�@mRNA�̏I�~�R�h���iUAG�AUAA�AUGA�j��A���ʂɂ���ƁA������q(RF-1,2,3)�����{�\�[����A���ʂɓ���C�I�~�R�h����F������BRF-1��UAA��UAG���CRF-2��UAA��UGA��F�����顂��̎��CRF-1, 2�͓���̃A�~�m�_�c��ŏI�~�R�h����F������i�y�v�`�h�A���`�R�h���Ƃ����j�B�Ȃ��CRF-3��GTP�����^���p�N���ŁCRF-1��RF-2�̃��{�\�[���ւ̌����𑣐i����
�@����ɂ��50S�T�u���j�b�g���y�v�`�W���g�����X�t�F���[�[������������CtRNA�Ɍ��������|���y�v�`�h�i�^���p�N���j��藣���B����Ń^���p�N�������͏I������B�Ȃ��C�T�v���b�T�[tRNA��RF���q��A���ʂŋ�������
�|���y�v�`�h���𗣂�����C���T�C�N�����q (RRF) �����{�\�[�������̂���̂���
 �@�^�j�����̃^���p�N������
�@�^�j�����̃^���p�N������
 �J�n���q�͂U�킠��F�@eIF-1a, eIF-2, eIF-3, eIF-4a/4b, eIF-4f, eIF-5
�J�n���q�͂U�킠��F�@eIF-1a, eIF-2, eIF-3, eIF-4a/4b, eIF-4f, eIF-5 �J�ntRNA (tRNAi)��Formyl-Met�ł͂Ȃ��A���`�I�j�����^�ԁB
�J�ntRNA (tRNAi)��Formyl-Met�ł͂Ȃ��A���`�I�j�����^�ԁB ���{�\�[���ւ�mRNA�̌����ł́C�L���b�v�\�����F�������B
���{�\�[���ւ�mRNA�̌����ł́C�L���b�v�\�����F�������B �J�n�����̌`���ɁAGTP�ȊO��ATP��K�v�Ƃ���B
�J�n�����̌`���ɁAGTP�ȊO��ATP��K�v�Ƃ���B �y�v�`�h���̐L���i�K�ɁA�������qeEF-1a�AeEF-1b�AeEF-2���֗^����B
�y�v�`�h���̐L���i�K�ɁA�������qeEF-1a�AeEF-1b�AeEF-2���֗^����B ������q�͂P��(eRF)�ŁAGTP��K�v�Ƃ���B
������q�͂P��(eRF)�ŁAGTP��K�v�Ƃ���B
|
 �@���j�����ɍ�p�������
�@���j�����ɍ�p�������| �R������ | ��@�@�@�p |
| �X�g���v�g�}�C�V�� �l�I�}�C�V�� �J�i�}�C�V�� �N�������t�F�j�R�[�� �e�g���T�C�N���� �G���X���}�C�V�� �s�����}�C�V�� �t�V�W���_ �J�X�K�}�C�V�� �����R�}�C�V�� �`�I�X�g���v�g�� �L���}�C�V�� �@ |
�J�nfMet-tRNA��P���ʂւ̌�����j�Q�B���{�\�[���ɂ�� ������|�����N�B ���� ���� �y�v�`�W���g�����X�t�F���[�[��j�Q�B �A�~�m�A�V��-tRNA�̌�����j�Q�B�����n�ȍ��I���B �V��50S���{�\�[���Ɍ������A�J�n�����̌`����j�Q�B �A�~�m�A�V��-tRNA�ގ��̂Ƃ��č�p�B EF-G�̗V����j�Q���A�~�m�A�V��-tRNA�̌�����j�Q�B �J�nfMet-tRNA��P���ʂւ̌�����j�Q�B �y�v�`�W���g�����X�t�F���[�[��j�Q�B EF-G��j�Q���ă��{�\�[���̓]�ʂ�j�Q�B EF-Tu�Ɍ������A�iEF-Tu-GTP-AAtRNA�j�����̂� ���{�\�[�������𑣐i�B |
 �@�^�j�����ɍ�p�������
�@�^�j�����ɍ�p�������| �j�Q�� | ��p |
| �A�u�����A���V�� �W�t�e���A�őf �N�������t�F�j�R�[�� �s�����}�C�V�� �t�V�W���_ �A�j�\�}�C�V�� �V�N���w�L�V�C�~�h �o�N�^�}�C�V�� �V���[�h�}�C�V�� �X�p���\�}�C�V�� �C���^�[�t�F���� |
�A�~�m�A�V��-tRNA�̌�����j�Q�B NAD��eIF-2�̔�����G�}���s���������q���B �~�g�R���h���A�̃y�v�`�W���g�����X�t�F���[�[��j�Q�B �A�~�m�A�V��-tRNA�̌�����j�Q�B�����n�ȍ��I���B eEF-2��j�Q���ă��{�\�[���̓]�ʂ�j�Q�B �y�v�`�W���g�����X�t�F���[�[��j�Q�B �y�v�`�h�]�ڂ�{�\�[���̓]�ʂ�j�Q�B �J�nMet-tRNA��P���ʂւ̌�����j�Q�B eIF-2-Met-tRNA-GTP�����̌`����j�Q�B ���{�\�[���̓]�ʂ�j�Q�B eIF-2�̃����_���ɂ��J�n��j�Q�B |
 |
�J�n���q3 �J�n���q1  mRNA |
 |
���j�����ɍ�p������̂�ԂŁC �^�j�����ɍ�p������̐� �����ɍ�p������̂�Ŏ���� |
|||
  |
�X�g���v�g�}�C�V�� �l�I�}�C�V�� �o�N�^�}�C�V�� �V���[�h�}�C�V�� |
�J�X�K�}�C�V�� �J�i�}�C�V�� �C���^�[�t�F���� |
||||
 30S �J�n������ |
||||||
 |
50S �T�u���j�b�g �G���X���}�C�V�� �G���X���}�C�V�� |
|||||
 70S �J�n������ |
�A�~�m�A�V��- tRNA�̌���  |
 |
 |
 |
||
 �e�g���T�C�N���� �s�����}�C�V�� |
||||||
| �A�u���� ���V��(risin) |
�y�v�`�h �]�� |
  |
�N�������t�F�j�R�[�� �����R�}�C�V�� �V�N���w�L�V�~�h �A�j�\�}�C�V�� |
|||
 |
 |
���{�\�[���� �]�� |
 |
|||
 |
 |
|||||
 �t�V�W���_ |
 �`�I�X�g���v�g�� �W�t�e���A�őf �V�N���w�L�V�~�h �X�p���\�}�C�V�� |
|||||
  
|
| �A���`�R�h�� ��1������ (5'���[) |
�Ή����� �R�h���� 3������ |
�R�h���� 3������ (3'���[) |
�Ή����� �A���`�R�h�� ��1������ |
| C | G | C | G, I |
| A | U | A | U, I |
| G | C, U | G | C, U |
| U | A, G | U | A, G, I |
| I | C, A, U |
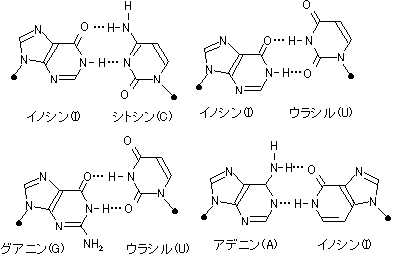
[��W���I�ȉ���̌`���i��炬�j]
  
|
| CAGUUGGGGC ACUGGGUGGG CGGCGGCGAC AGCGGCGCCA CGCGCAGGCU GGAGGCCGCC�@��P�J�n�R�h���iCUG) GAGGCUCGCC AUGCCGGGAG AACUCUAACU CCCCCAUGGA GUCGGCCGAC UUCUACGAGG�@��Q�C��R�J�n�R�h�� |
| UGGAGCCGCG GCCCCCGAUG AGCAGUCACC UCCAGAGCCC CCCGCACGCG CCCAGCAACG�@��S�J�n�R�h�� CCCGCCUUUG GCUUUCCCCG GGGCGCGGGC CCCGCGCCGC CCCCAGCCCC ACCUGCCGCC CCGGAGCCGC UGGGCGGAUC UGCGAGCACG AGACGUCUAU AGACAUCAGC GCCUACAUCG ACCCGGCCGC CUUCAACGAC GAGUUCCUGG CCGACCUCUU CCAGCACAGC CGACAGCAGG AGAAGGCCAA GGCGGCGGCG GGCCCCGCGG GUGGCGGCGG UGACUUUGAC UACCCGGGAG |
| CCCCGGCGGG CCCCGGCGGC GCGGUCAUGU CCGCGGGGGC GCACGGGCCC CCUCCCGGCU�@��T�J�n�R�h�� |
| ACGGCUGUGC GGCGGCCGGC UACCUGGACG GCAGGCUGGA GCCCCUGUAC GAGCGCGUCG GGGCGCCCGC GCUACGGCCG CUGGUGAUCA AACAAGAGCC CCGCGAGGAG GACGAGGCGA AGCAGCUGGC GCUGGCCGGC CUCUUCCCCU ACCAGCCACC GCCGCCACCG CCACCGCCGC ACCCGCACGC GUCUCCCGCG CACCUGGCCG CCCCCCACUU GCAGUUCCAG AUCGCGCACU GCGGCCAGAC CACCAUGCAC CUACAGCCUG GCCACCCCAC ACCGCCGCCC ACGCCCGUGC CCAGCCCGCA CGCUGCGCCC GCCUUGGGUG CUGCGGGCCU GCCUGGCCCC GGGAGCGCGC UCAAGGGCUU GGCCGGUGCG CACCCCGACC UCCGCACGGG AGGCGGCGGC GGUGGCAGCG GUGCCGGUGC GGGCAAAGCC AAGAAGUCGG UGGACAAGAA CAGCAACGAG UACCGGGUAC GGCGGGAACG CAACAACAUC GCGGUGCGCA AGAGCCGAGA UAAAGCCAAA CAACGCAACG UGGAGACGCA ACAGAAGGUG CUGGAGUUGA CCAGUGACAA UGACCGCCUG CGCAAGCGGG UGGAACAGCU GAGCCGUGAA CUGGACACGC UGCGGGGCAU CUUCCGCCAG CUGCCUGAGA GCUCCUUGGU CAAGGCCAUG GCAACUGCGC GUGAGGCGCG CGGCUGCGGG ACCGCCUUGG GCCGGCCCCC UGGCUGGAGA CCCAGAGGAU GGUUUCGGGU CGCUGGAUCU CUAGGCUGCC |
| CGGGCCGCGC AAGCCAGGAC UAGgagauuc cgguguggcc ugaaagccug gccugcuccg�@�I�~�R�h��(UAG) |
  
|
�@�|��ɂ������O�I�Ȏ���Ƃ��āC�ǂݎ��g�̈ړ��C���{�\�[���̒���C�I�~�R�h���̓ǂݔ���i���[�h�X���[�j�C�I�~�R�h��UGA�̓Ǒւ��i�Z���m�V�X�e�C��[NH2-CH(CH2-SeH)-COOH]�Ƃ��Ė|��j�Ȃǂ���B������mRNA�ɂ����`�Í��̏�������(recoding)�Ƃ����B����CRNA�G�f�B�e�B���O�̂悤�ɁC��RNA�̒i�K�ő啝�ȕύX���s����ꍇ������B
 ���[�h�X���[(read through)
���[�h�X���[(read through)
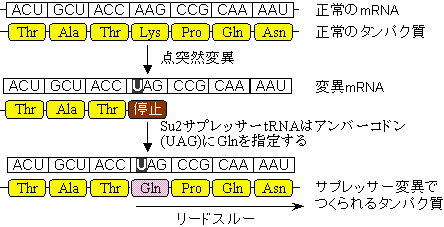
[�ψ�tRNA�i�T�v���b�T�[tRNA�j�ɂ�郊�[�h�X���[]
| �y���g���E�B���X�̃��[�h�X���[�z �@���g���E�B���X��gag�i�k�^���p�N����`�q�j��pol�i�t�]�ʍy�f��`�q�j�̊Ԃɂ͏I�~�R�h�����P����B�ʏ�́AGag�^���p�N���������Ă��邪�A���ʂ̃T�v���b�T�[tRNA���I�~�R�h�������[�h�X���[���A���ʂ�Gag-Pol�Z���^���p�N����������B���̗Z���^���p�N���̌��蕪���ɂ��A���n�^��Pol�^���p�N����������B |
  
|
 �y�v�`�h�����̌��蕪���cN�[Met�̏����AN�[�V�O�i���y�v�`�h�̏����A���蕪���ɂ��y�f�O���(zymogen)�̊������ȂǁB
�y�v�`�h�����̌��蕪���cN�[Met�̏����AN�[�V�O�i���y�v�`�h�̏����A���蕪���ɂ��y�f�O���(zymogen)�̊������ȂǁB ��y�f�A�����C�I���A�w���Ȃǂ̕t��
��y�f�A�����C�I���A�w���Ȃǂ̕t�� �V���y�����ɂ�鍂���\���̌`��
�V���y�����ɂ�鍂���\���̌`�� �y�v�`�h�����̃V�X��(�����Pro�c��)
�y�v�`�h�����̃V�X��(�����Pro�c��) �W�X���t�B�h�����̌`��
�W�X���t�B�h�����̌`�� �A�~�m�_�����̏C���c�����_���A���`�����A�A�V�����A�q�h���L�V�����A�����̕t���AGlu�̃J���{�L�V�����ALys�c��Ԃ̉ˋ��A�|��ADP���{�V�����A���r�L�`�����ȂǁB
�A�~�m�_�����̏C���c�����_���A���`�����A�A�V�����A�q�h���L�V�����A�����̕t���AGlu�̃J���{�L�V�����ALys�c��Ԃ̉ˋ��A�|��ADP���{�V�����A���r�L�`�����ȂǁB ����̃A�~�m�_��D�^���i�܂�Ȏ��ہj
����̃A�~�m�_��D�^���i�܂�Ȏ��ہj �^���p�N���̎��ȃX�v���C�V���O�i�܂�Ȏ��ہj
�^���p�N���̎��ȃX�v���C�V���O�i�܂�Ȏ��ہj
[�^���p�N���X�v���C�V���O]
�藣����镔�����C���e�C��(intein)�A�A������镔�����G�N�X�e�C��(extein)�Ƃ����B�@RNA��intron, exon��^�������O�ł���B
 �^���p�N���̃\�[�e�B���O
�^���p�N���̃\�[�e�B���O
�@1) ���E�̖���ʉ߂���^���p�N���́AN�[�ɃV�O�i���y�v�`�h�����B
�@2) ���E�̓��o�ɗ��܂�^���p�N���́AC�[��Lys-Asp-Glu-Leu�z������B
�@3) �j�Ɉڍs����^���p�N���ɂ�N�[�Ɋj�ڍs�V�O�i���z��iLys��Arg��5�c��j������B
�@4) �~�g�R���h���A��t�Α̂ɍs���^���p�N����N�[��20�c��قǂ̓����I�Ȕz����B
�@5) �y���I�L�V�\�[���ɍs���^���p�N����C�[��Ser-Lys-Leu�����B
 (molecular mimicry) (molecular mimicry) |
 |
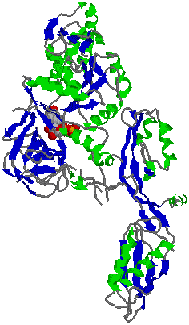 |
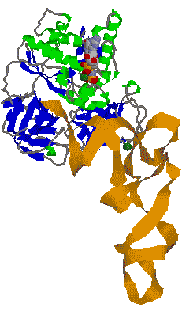 tRNA���� tRNA���� |
| [���q�[�Ԃ̗�] | |
| �i���j�������qG�C�i�E�j�A�~�m�A�V��-tRNA-EF-Tu -GTP�����́B �^���p�N��������ŁCtRNA������Ŏ����B tRNA�ƌ������Ă��Ȃ��Ă�EF-G�����{�\�[����A���ʂɌ����ł���̂́C ���̂悤�ȗ��̍\�����Ƃ邽�߂ł��낤� |
|

|